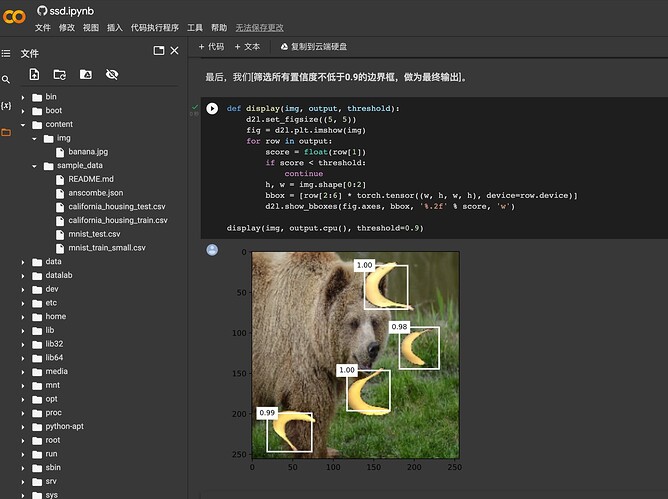
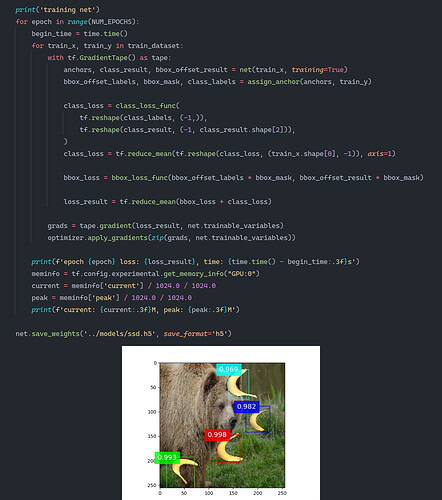
RuntimeError: [Errno 2] No such file or directory: ‘…/img/banana.jpg’,大家有遇到过这种错误吗
你电脑上是没有这个banana.jpg的,我们可以从数据集中随便选一张测试集的图片作为样本。
譬如:
X = tv.io.read_image("../dataset/banana-detection/bananas_val/images/10.png").unsqueeze(0).float()
cdn出了问题,js加载不出来,建议用加速器加速一下(挂梯子)。
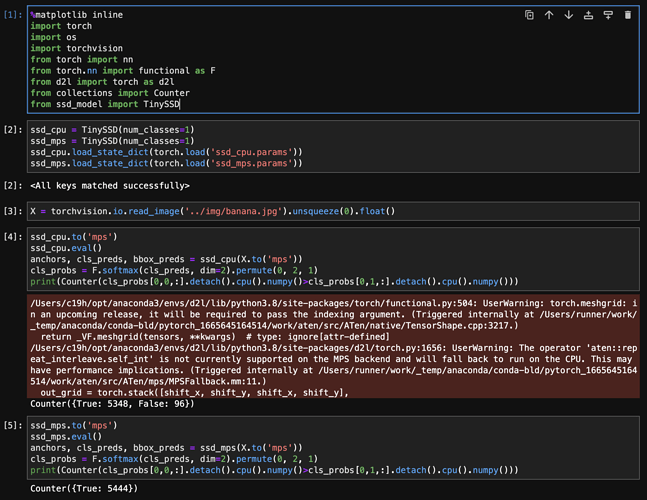
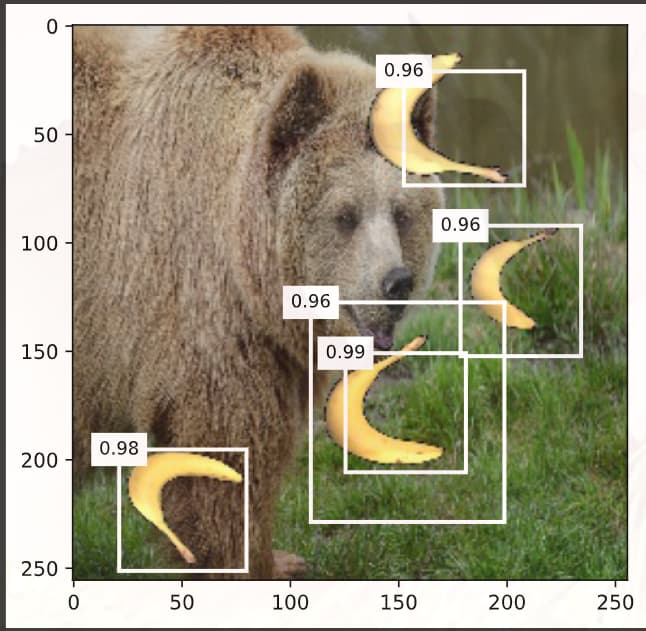
本人用的是mac M1,分别在device='cpu’和device='mps’上训练了两个TinySSD模型(除了device参数不一样,其他都相同)。用课程中的banana.jpg测试模型得出cpu上的模型能正常识别锚框中的香蕉类,而mps模型则全识别为背景类,这是何原因?
我觉得是调参调出来的,这样设置检测的效果最好。
我自己也试过不同的锚框大小,结果很差。通过debug发现,如果锚框大小和真实的bbox相差较大,那么在锚框和bbox对应的函数(assign_anchor_to_bbox)中,只有一个anchor能被分配给bbox(在本案例中,一张图片里面只有一个香蕉bbox),导致正负样本差距特别大(我的情况是1:5444)。这会导致所有的anchor都被分类为背景,进而导致anchor的偏移值收敛效果不好。
如果就按照教程来设置anchor大小,那么正负样本的比例就可以达到32:5444左右,结果也就正常了。
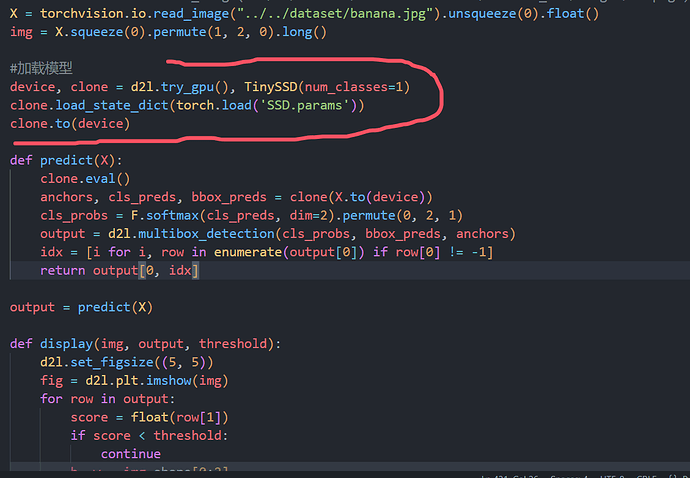
如果在测试代码的过程中,提示 tensor 分布在两个设备上的提示错误,可以按照下面的方式来进行纠正。
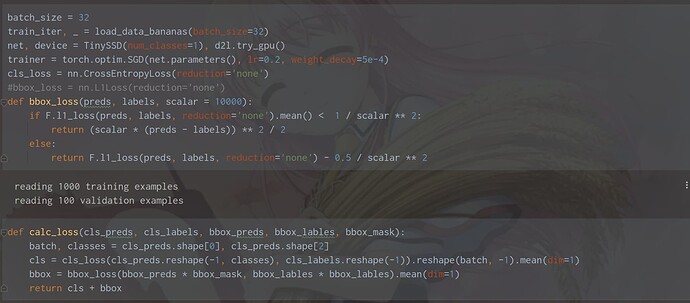
# model training
num_epochs , timer= 20, d2l.Timer()
animator = d2l.Animator(xlabel = 'epoch' ,xlim = [1,num_epochs],
legend =['classes error','bbox mae'])
net = net.to(device)
for epoch in range(num_epochs):
metric = d2l.Accumulator(4)
net.train()
for features ,target in train_iter :
timer.start()
trainer.zero_grad()
X, Y = features.to(device), target.to(device)
# X ,Y = features, target
anchors,cls_pred , bbox_preds = net(X)
#
bbox_labels,bbox_masks,cls_labels = d2l.multibox_target(anchors,Y)
#
bbox_labels, bbox_masks,cls_labels = bbox_labels.to(device),bbox_masks.to(device),cls_labels.to(device )
cls_preds = cls_pred.to(device)
l = calc_loss(cls_preds, cls_labels,bbox_preds ,bbox_labels,
bbox_masks)
# l.to(device)
l.mean().backward()
trainer.step()
metric.add(cls_eval(cls_preds, cls_labels),cls_labels.numel(),
bbox_eval(bbox_preds,bbox_labels,bbox_masks),
bbox_labels.numel())
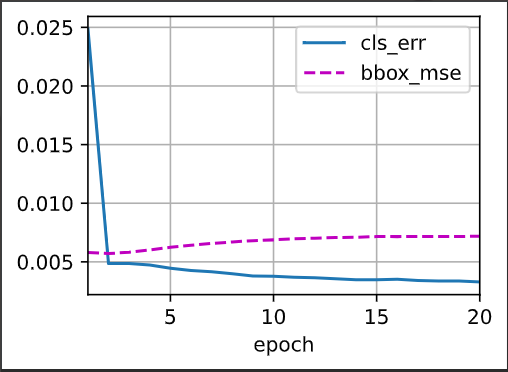
cls_err,bbox_mae = 1 - metric[0] / metric[1] ,metric[2]/metric[3]
animator.add(epoch + 1 , (cls_err , bbox_mae))
print(f'class err { cls_err :.2e}, bbox_mae {bbox_mae:.2e}')
print(f'{len(train_iter.dataset)/ time.stop():.1f} examples/sec on ',f'{str(device)}')
修改的地方主要是把在计算损失的时候,把对应的数据移动到了cuda 上进行操作
这里使用d2l.load_data_bananas加载的数据是不是没归一化啊
mps最后误差会很大,所以识别不好,我也是这样,估计是mps里的数位不同导致精度的问题吗?我看是long()那里
batch_size = 32
train_iter, _ = d2l.load_data_bananas(batch_size)这一步报错BadZipFile: File is not a zip file怎么办啊
有人尝试过将高宽减半块换为带残差的高宽减半吗?我将之前残差单元根据这个高宽减半块改了填充和步幅还有核大小,但是输出结果的bboxmae一开始就是一个接近0的值并且几乎不变